Systems and Synthetic Biology Laboratory
School of Chemical and Biological Engineering
Seoul National University
1 Gwanak-ro, Gwanak-gu, Seoul, 08826, Republic of Korea
Tel: 02-880-2274
E-mail: swseo@snu.ac.kr

포스텍 화학공학과 학사
포스텍 화학공학과 박사
| 기간 | 기관명 | 직위 및 직급 |
|---|---|---|
| 2012.2 – 2013.2 | 포스텍 화학공학과 | Postdoc 연구원 |
| 2013.3 – 2015.12 | Department of Bioengineering, University of California San Diego (UCSD) | Postdoc 연구원 |
| 2016.3 – 현재 | 서울대학교 화학생물공학부 | 조교수 |

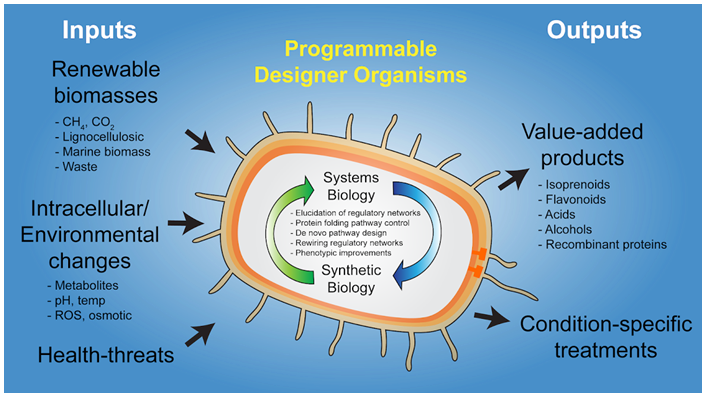
본 연구실은 1) 융합 오믹스 분석을 바탕으로 생체 시스템의 조절 네트워크를 이해하고, 2) 미생물을 효과적으로 개량할 수 있도록 유전자 재조합을 위한 합성생물학적 분자 도구들을 개발함으로써, 3) 확보한 생물학적 정보를 기반으로 합성생물학적 도구들을 활용하여 특정한 목적에 맞도록 프로그래밍 된 인공 미생물을 개발하는 연구를 수행한다.
DNA 서열은 생명체에 대한 많은 정보를 담고 있기 때문에 이를 분석하면 대사회로를 파악하고 조작할 수 있다. 2005년 차세대 염기서열 분석 기술이 개발되면서 현재는 미생물의 유전체를 단시간에 분석할 수 있으며 이를 통해 신규 미생물의 유전정보 파악, 진화 개체의 변이 위치 분석이 가능하다. 또한, 전사체, 전사개시위치, 번역체, 상호작용체 등 목적에 맞게 준비된 염기서열을 분석함으로써 더욱 많은 정보를 얻고 있다.
전사체는 특정 조건 및 환경에서 발현된 세포 내 RNA의 총체이다. 세포로부터 전사체를 추출 한 뒤 단백질 서열을 암호화하는 mRNA만을 골라 시퀀싱하면 세포의 유전정보 및 환경변화에 따라 달라지는 유전자 발현 수준에 대한 정보, 유전자 발현 단위 구조를 파악할 수 있으며 이를 전사체 분석이라고 한다. 전사개시위치 분석은 mRNA의 전사가 시작된 위치만을 집중적으로 시퀀싱 함으로써 전사개시위치를 1bp 단위로 파악 할 수 있으며 이를 통해 프로모터 서열을 도출할 수 있다.
전사체로부터 분해되거나 전사 후 공정을 거쳐서 만들어진 5'말단을 가진 RNA를 제거하고(TEX 처리) 전사 시작 위치염기의 triphosphate를 monophosphate로 바꿔주면 (TAP 처리) 시퀀싱 라이브러리 증폭을 위한 어답터가 붙을 수 있고 이를 이용해 시퀀싱을 진행하여 전사체 분석 결과와의 비교분석을 통해 전사개시위치를 파악할 수 있다.
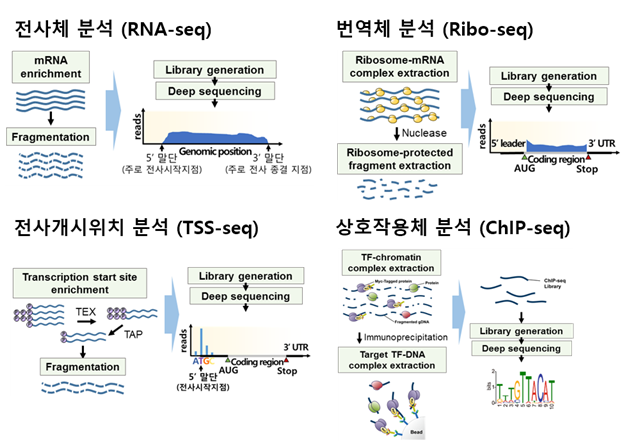
많은 경우에 있어서 실제 생명 현상을 일으키는 것은 단백질이기 때문에 단백체에 대한 연구도 진행되어 왔다. 그러나 기술의 한계로 단백질 서열 분석은 전체 유전체에 대한 접근이 어렵다. 본 연구실은 세포내 번역 발현량에 대한 정보를 얻기 위해 번역체 분석을 활용하고 있다. 번역체 분석은 세포내 mRNA를 결합한 리보솜이 떨어지지 않도록 추출한 후 뉴클레아제 처리를 통해 리보솜이 보호하고 있지 않은 부분은 제거한 뒤 보호된 영역의 서열을 분석하는 기술이다. 이러한 결과를 전사체 분석 결과와 비교하면 정확한 번역 단위 구조를 알 수 있고, 단일 mRNA를 기준으로 하는 번역 발현량을 알 수 있다. 이를 통해 5'UTR 서열과 번역 효율의 관계를 분석할 수 있다. 또한 한 유전자 내에서의 리보솜 정체 구간/밀집구간 등 번역 프로파일링이 가능하다.
세포내에는 다양한 DNA에 결합하는 다양한 단백질들이 존재하며, 이 중 전사조절인자의 결합 위치를 파악하면 전사조절 네트워크를 발굴할 수 있다. 목표가 되는 전자조절인자에 표지를 달아준 세포를 배양하여 포름알데히드를 처리하면 단백질과 DNA가 단단하게 결합하게 되고, 세포로부터 염색체를 추출하여 표지에 특이적 항체를 사용하여 전사조절인자-DNA 복합체를 침강시킬 수 있다. 회수된 서열을 분석하여 전사조절인자가 결합하는 모티프를 1 bp 단위로 정확하게 파악할 수 있다. 상호작용체 분석결과와 전사체 분석 결과를 종합하여 해석하면 전사조절인자의 레귤론을 발굴할 수 있다. 본 연구실은 미생물의 대사회로 정보 및 환경변화에 따른 조절기작을 파악하기 위해 이러한 실험적 기술을 연구실 안에서 구현하고 새로운 기술 개발을 위한 연구를 한다.
2. 합성생물학 기반 분자조절 도구 개발 및 균주 개량 연구생명현상에는 DNA, RNA, 단백질로 이어지는 생명체의 중심원리가 중요하게 작용하며, 미생물을 개량하기 위해서는 이러한 단계를 조작하여 유전자의 발현량을 정교하게 예측하고 제어할 수 있어야 한다. 본 연구실에서는 합성생물학에서 필요로 하는 정량화된 발현 조절 부품 및 기존의 부품들을 조립하여 복잡한 명령을 수행할 수 있는 생체 시스템을 구축하는 것을 목표로 한다. 또한 이러한 조절 기술을 활용하여 미생물을 개량해 다양한 생리활성 물질을 생산하는 연구를 수행한다.
분자생물학적 기술의 발달로 유전자 조작이 용이해진 대장균은 균주개량의 모델 시스템으로 활용되어왔다. 최근에는 서로 다른 미생물의 특징에 주목하여 목적에 맞는 미생물을 개량하고자 하는 시도가 이뤄지고 있다. 본 연구실에서는 대장균 외에 메탄자화균(Methanotroph), 해양유래 미생물, 유산균(Lactic acid bacteria), 유지효모(Yarrowia lipolytica)를 개량하여 여러 원료로부터 화합물을 생산하고자 한다. 이러한 다양한 미생물들이 가지는 장점들에도 불구하고 비 전통적인 미생물들을 개량하는데 있어 유전체에 대한 정보 부족, 적절한 유전자 조작 방법 부족 등의 제약을 갖고 있다. 따라서 본 연구실에서는 비 전통적 균주들의 유전체, 전사체, 번역체, 상호작용체 등 다양한 오믹스 분석을 통하여 미생물의 대사 네트워크를 파악하고 프로모터, UTR 등 유전자 발현 부품들을 발굴하는 연구를 진행하고 있다. 이러한 연구를 통해 각 균주의 장점을 극대화한 균주 개량 전략을 수립할 수 있을 것이며, 융합 오믹스 분석을 통해 확보한 생물학적 부품을 이용한 대사경로 최적화 설계를 통해 경제적인 화합물 생산이 가능할 것으로 전망한다.