Department of Chemical Engineering, School of Energy and Chemical Engineering, UNIST
50 UNIST-gil, Ulsan 44919, Republic of Korea

Ph.D., Bioengineering, University of California, San Diego, California, USA
B.S., Computer Science and Engineering & Biological Sciences, Seoul National University
| 기간 | 기관명 | 직위 및 직급 |
|---|---|---|
| 2020.09~present | UNIST | Associate Professor |
| 2018.07~2020.08 | UNIST | Assistant Professor |
| 2016.03~2018.06 | Kyung Hee University | Assistant Professor |

시스템생물학은 유기체의 생명현상을 시스템적으로 규명하고 생명체와 환경 간의 연관성을 분석하는 연구 분야로, 전사체 분석과 같은 실험 방법을 통해 얻은 데이터를 기반으로 전사 조절 네트워크를 규명하는 연구를 하고 있습니다. 또한, 전사체 데이터를 활용하여 대사 모델(Genome-scale metabolic models, GEMs)를 구축하여 실험적으로 규명하기 힘든 다량의 유전체 변화를 예측하는 연구를 진행하고 있습니다. 그뿐만 아니라 기계학습을 생물/화학적 데이터에 활용하여 단백질 구조 예측 및 공정 개선과 같은 연구를 수행하고 있습니다. 이를 통해 생명 중심의 연구에서 화학공학, 그리고 기계학습 쪽으로 점차 연구 분야를 넓혀가고 있습니다.
본 연구실은 균주의 전사 조절을 분석하기 위해 차세대 염기서열 분석법 (Next Generation Sequencing, NGS)을 활용하는 전사체 분석 (RNA-seq)과 크로마틴 면역침강법 (ChIP-exo)을 진행하고 있습니다. 또한, 실험데이터의 분석을 위해 자체 제작한 분석 파이프라인과 차세대 염기서열 분석법 데이터 시각화 프로그램 소프트웨어 (Metascope)를 활용하여 여러 가지 실험 결과를 통합하여 전사인자에 대한 포괄적인 정보를 분석하고 있습니다.
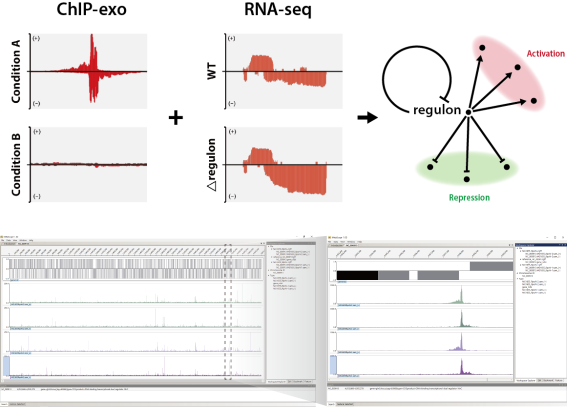 차세대 염기서열 분석법을 활용한 전사 조절 연구의 개요와 분석 시각화 도구인 Metascope
차세대 염기서열 분석법을 활용한 전사 조절 연구의 개요와 분석 시각화 도구인 Metascope
고 처리량 시퀀싱 기술로 읽어진 고품질의 유전체 서열을 기반으로 다양한 데이터베이스를 통해 각 유전자의 주석을 파악함으로써 유전체의 기본 정보를 확보하고 있습니다. 이와 관련된 여러 대사 물질 및 화학 반응을 확인하여 선형 모델링을 기반으로 대사 네트워크를 구성하여 이를 연구에 활용하고 있습니다. 이는 목표 물질에 관련된 생합성 경로를 확인하고 시뮬레이션 함으로써 대사 흐름을 예측할 수 있으며 또한 유전자 제거 혹은 유전자 삽입과 같은 조작을 간단히 진행할 수 있으므로 새로운 생합성 경로를 발굴하는 방법에도 유용하게 사용될 수 있습니다.
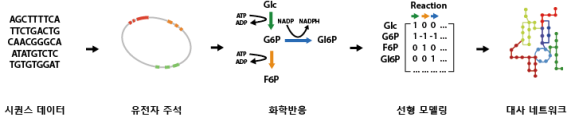 선형 모델링을 활용한 대사 네트워크 구축 및 연구
선형 모델링을 활용한 대사 네트워크 구축 및 연구
C1 가스 전환 박테리아 중 Methylorubrum extorquens AM1은 산업용으로 주목받고 있지만 아직 균주 자체에 관한 연구가 많이 진행되지는 않은 상태입니다. 본 연구실에서는 이 균주의 유전체를 기반으로 하여 시스템 생물학적 방법을 통해 균주의 특성을 파악하는 연구를 진행하고 있습니다. 이를 위하여 앞서 언급한 전사 조절 연구와 함께 전사체 분석, 대사 모델 구축과 같은 방법을 사용하고 있고, 이를 통해 M. extorquens AM1 균주의 기능 유전체학적 지도를 완성하여 목표로 하는 생물 전환을 효율적으로 이루어낼 수 있을 것으로 예상합니다.
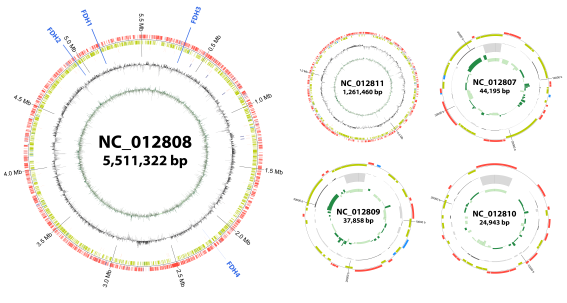 M. extorquens AM1 유전체 지도
M. extorquens AM1 유전체 지도
기계학습을 활용한 단백질 구조 예측 프로그램인 알파폴드가 (AlphaFold) 공개되면서 수많은 규명되지 않은 단백질 구조들이 공개되고 있습니다. 이를 활용하여 효소 돌연변이의 기능예측과 같은 연구를 진행하고 있으며, 궁극적으로 보조인자 대체 효소 설계와 같은 연구를 목표로 하고 있습니다.
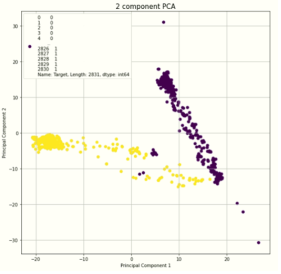 보조인자에 따른 효소 클러스터 분석
보조인자에 따른 효소 클러스터 분석
베이지안 최적화와 생성적 적대 신경망과 같은 여러 가지 방법을 활용하여 배터리 및 화학 반응에 적용하는 연구를 진행하고 있습니다. 선형판별분석(LDA, Linear Discriminant Analysis) 를 통해 upconversion nanoparticle 촉각센서 필체감정시스템을 구현하였으며, 이를 통해 새로운 필체 분석 방식을 제안함과 동시에, high spatial resolution 을 특징으로 하는 촉각센서의 경쟁력을 여러 분야에 적용할 수 있는 가능성을 확인하였습니다.
 구현된 실시간 점자 인식 시스템
구현된 실시간 점자 인식 시스템